Nat Commun:从循环细胞游离DNA甲基化中破译肿瘤片段,用于癌症早期诊断
时间:2023-01-03 15:00:56 热度:37.1℃ 作者:网络
肿瘤来源的循环细胞游离DNA (cfDNA)为癌症早期诊断提供了重要线索,但其敏感性往往较低。
2022年12月13日,清华大学刘民,广东省第二人民医院程伟彬及田军章共同通讯(清华大学为第一单位)在Nature Communications 发表题为“Tumor fractions deciphered from circulating cell-free DNA methylation for cancer early diagnosis”的研究论文,该研究提出了一种癌症早期诊断方法,使用从循环cfDNA甲基化特征中破译的肿瘤片段。在两个独立的数据集中,随着癌症的进展,来自癌症患者的肿瘤来源的cfDNA的估计分数显著增加。
利用预测的肿瘤部分,该研究建立了一个贝叶斯诊断模型,其中训练样本仅来自晚期患者和健康个体。当在早期患者和健康个体上验证时,该模型对癌症早期检测的敏感性为86.1%,对肿瘤定位的平均准确性为76.9%,特异性为94.7%。通过强调肿瘤部分在癌症早期诊断中的潜力,该研究方法可以进一步应用于癌症筛查和肿瘤进展监测。

尽管最近癌症治疗取得了进展,但早期诊断已被最终证明可以提高患者的生存机会,甚至为临床医生提供了通过手术切除肿瘤来治愈癌症的机会。然而,临床癌症筛查仍依赖于非分子技术,且特异性和敏感性均较低。由于缺乏有效的癌症筛查方法,大多数癌症患者诊断较晚,从而错过了理想的治疗期。作为一种液体活检分析物,外周血血浆中的循环无细胞DNA (cfDNA)由于其非侵入性特性已成为一种有前途的癌症早期诊断生物标志物。然而,肿瘤源性cfDNA相对于从癌症患者中提取的总血浆cfDNA的比例还不足以进行常规诊断。
为了精确评估肿瘤来源的cfDNA,拷贝数变异(CNVs)特异性突变和甲基化谱被广泛利用作为cfDNA的鉴别分子特征。基于CNVs的早期肿瘤片段预测方法依赖于成本高昂的全基因组测序(WGS),其序列覆盖率约为100倍。最先进的方法ichorCNA和ACE是从低覆盖率WGS发展而来的,用于量化cfDNA中的肿瘤部分。然而,由于缺乏足够的非整倍性和染色体不稳定性,这两种方法都可能无法提供肿瘤分数的可靠估计。尽管cfDNA中的肿瘤源性突变也可以用来区分潜在的癌症患者和正常对照组,但它们仍然难以检测,因为突变取决于肿瘤类型、癌症分期、生物噪声和技术敏感性。此外,基于突变的诊断方法在定位肿瘤的组织起源(TOO)方面存在很大困难,因为多种恶性肿瘤类型共享许多驱动突变。
相比之下,作为一种重要的表观遗传修饰,DNA甲基化特征在健康个体与各种疾病特别是恶性肿瘤患者之间表现出显著差异。因此,最近的研究分析了用于癌症诊断的区分健康个体与恶性肿瘤患者的差异甲基化探针或区域(DMPs/DMRs)。由于循环中的cfDNA有多种来源,每个CpG位点的甲基化水平本质上是源自血细胞和多个组织的混合信号,包括产生与肿瘤发生相关的cfDNA的组织。因此,通过反卷积混合甲基化标记来估计cfDNA的TOO是可行的,这可能使预测原发肿瘤的位置成为可能。
在这项工作中提出了一种癌症早期诊断方法,使用cfDNA甲基化谱来破译肿瘤片段并定位潜在的肿瘤。提出了一种半无参考反褶褶(SRFD)算法来自动从cfDNA甲基化签名中学习参考数据库,而不是从组织数据中手动连接参考数据库。利用这一开发的策略,观察到在包括多种癌症类型的两个独立患者血浆数据集中,随着癌症进展,估计的肿瘤部分显著增长(p值< 0.005)。
利用肿瘤分数和机器学习分类器模型的优点,建立了一个贝叶斯诊断模型,命名为SRFD-Bayes,以做出最终的诊断决策,并在模拟中优于基于分类器的诊断模型。由于晚期癌症患者在临床实践中比早期患者更常见,研究者检查了晚期肿瘤患者来构建我们的诊断模型,并使用早期患者的样本验证了该模型。该研究方法对癌症早期检测的敏感性为86.1%,特异性为94.7%,优于先前的模型。
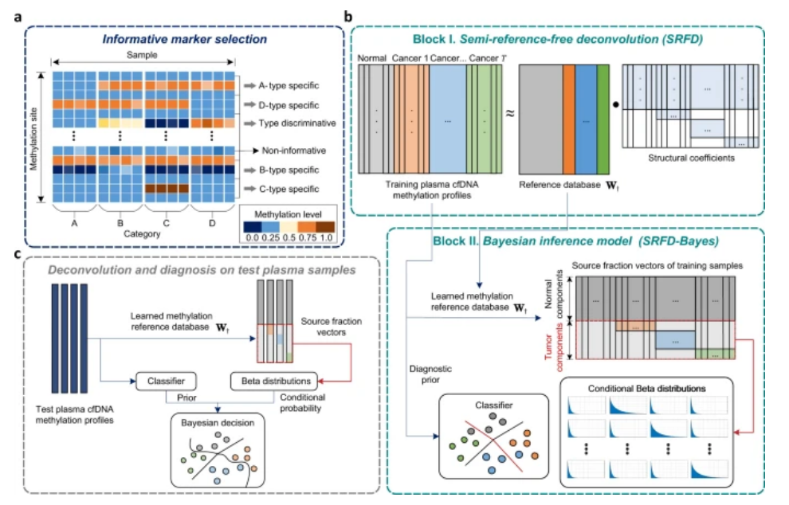
癌症早期诊断方法概述(图源自Nature Communications )
综上所述,该研究提供了一种可用于监测肿瘤进展的有效工具,在大规模癌症筛查中具有很大的潜力。
参考消息:
https://doi.org/10.1038/s41467-022-35320-3


